dplyrのmutateってちょっと遅い?
{dplyr}が1.0.0になってacrossが追加されて、*_atや*_all、*_eachがじゃなくてもacrossでやる風潮(?)になって、(もう1.0.2だけど今更)便利だなぁと{dplyr}で遊んでいたら(前々から思っていたけど)*_eachが実は体感できるレベルで一瞬待たされることに気づいてしまった……
そこで、どの程{dplyr}がどの程度の速度なのかちょっとみてみた。今回は列変化を{base}と{dplyr}のmutateをつかって比べてみた。昔の{plyr}に比べて{dplyr}は早くなっているとか、{dplyr}はやっぱり遅いよねという話はたまに見かけるけど、どの程度遅いのかみてみた。
結論としては条件によるかもしれないけど、{dplyr}は遅かった。主語が{dplyr}だとさすがにでかすぎるのでmutateが遅い程度にしておく。基本的にただの与太話。
ただし書きやすさは別。統一された思想の{tidyverse}は書きやすい。
計測条件
100行3列の0と1からなるデータフレームをsampleを使って作って、3列すべてを理論型に変換するというものを1000回やった時間で計測してみる。
時間の計測にはsystem.timeをつかった。
{base}でやってみる
単純に{base}のas.logicalをつかってそれだけでやってみる。
system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat$x <- as.logical(dat$x) dat$y <- as.logical(dat$y) dat$z <- as.logical(dat$z) } )
## user system elapsed
## 0.57 0.05 0.62
圧倒的に早い。他のコードでも、理論型への変換にはas.logicalを使っているのでこれより早くはなりえない?
次に、{base}時代の列に関数適用して変換してやる定番だったtransformをつかってやってみる。
system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- transform(dat, x = as.logical(x), y = as.logical(y), z = as.logical(z)) } )
## user system elapsed
## 0.75 0.03 0.80
これもあまぁまぁ早い。
次に、ちょっと変化球でapplyつかってやってみる。applyは行列で返すので同じ条件で結果が出力されるようにdata.frameで結果を調整している。
system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- data.frame(apply(dat, 2, as.logical)) } )
## user system elapsed
## 0.89 0.01 0.91
もう一回data.frame関数使っているのにそんなに遅くない。 結構早い。
{dplyr}のmutateを使ってみる
最初に{dplyr}をlibraryで呼び出しておく。
library("dplyr")
##
## Attaching package: 'dplyr'
## The following objects are masked from 'package:stats':
##
## filter, lag
## The following objects are masked from 'package:base':
##
## intersect, setdiff, setequal, union
一番単純にmutateで書いてみる。
system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- mutate(dat, x = as.logical(x), y = as.logical(y), z = as.logical(z)) } )
## user system elapsed
## 3.69 0.05 3.77
急に遅くなる。
acrossの登場によりsupersededになったmutate_all、mutate_atを使ってみる。
system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- mutate_all(dat, as.logical) } )
## user system elapsed
## 4.53 0.06 4.64
system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- mutate_at(dat, vars(x, y, z), as.logical) } )
## user system elapsed
## 8.24 0.06 8.41
さらにちょっと遅い。
結構前(0.7.0)からdeprecatedになったmutate_eachをつかってみる。
system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- mutate_each(dat, as.logical, x, y, z) } )
## Warning: `mutate_each_()` is deprecated as of dplyr 0.7.0.
## Please use `across()` instead.
## This warning is displayed once every 8 hours.
## Call `lifecycle::last_warnings()` to see where this warning was generated.
## user system elapsed
## 15.38 0.19 16.22
圧倒的に遅い。一連の中で最遅。そして、mutate_atとmutate_eachで適応させる関数と適応させる列の選択の順番がちがって時々間違える罠がある。
1.0.0で登場したacrossをつかってみる。everythingを使うとmutate_allっぽく、行を選択して入れるとmutate_atやmutate_eachっぽくなる。いい。
system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- mutate(dat, across(everything(), as.logical)) } )
## user system elapsed
## 6.30 0.13 6.61
system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- mutate(dat, across(c(x,y,z), as.logical)) } )
## user system elapsed
## 7.75 0.11 8.05
everythingも列指定もほぼ一緒、mutate_eachよりも早い。
結論
- 圧倒的に
{base}で書いたほうが早い。 {dplyr}のmutateは使う関数によって結構速度に差があるが、基本的に{base}で書くより遅い。順番的はmutate<mutate_all<mutate+acrossormutate_at\<<mutate_eachになっている。
もっと大きなデータだったりすると様相が違うかもしれないけどこんな感じだった。
おまけ
単回じゃなく、10回ずつやった結果の箱ひげ図を作ってみる。
1000回のシミュレート×10回×8種類の関数で正味8万回も計算してみる。
library(tidyverse)
## -- Attaching packages ----------------- tidyverse 1.3.0 --
## √ ggplot2 3.3.2 √ purrr 0.3.4
## √ tibble 3.0.3 √ dplyr 1.0.2
## √ tidyr 1.1.2 √ stringr 1.4.0
## √ readr 1.3.1 √ forcats 0.5.0
## -- Conflicts -------------------- tidyverse_conflicts() --
## x dplyr::filter() masks stats::filter()
## x dplyr::lag() masks stats::lag()
func_mutate <- NULL func_mutate_across <- NULL func_mutate_all <- NULL func_mutate_at <- NULL func_mutate_each <- NULL func_as.logical <- NULL func_transform <- NULL func_apply <- NULL for(j in 1:10){ func_mutate[j] <- system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- mutate(dat, x = as.logical(x), y = as.logical(y), z = as.logical(z)) } ) func_mutate_across[j] <- system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- mutate(dat, across(c(x,y,z), as.logical)) } ) func_mutate_all[j] <- system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- mutate_all(dat, as.logical) } ) func_mutate_at[j] <- system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- mutate_at(dat, vars(x, y, z), as.logical) } ) func_mutate_each[j] <- system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- mutate_each(dat, as.logical, x, y, z) } ) func_as.logical[j] <- system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat$x <- as.logical(dat$x) dat$y <- as.logical(dat$y) dat$z <- as.logical(dat$z) } ) func_transform[j] <- system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- transform(dat, x = as.logical(x), y = as.logical(y), z = as.logical(z)) } ) func_apply[j] <- system.time( for(i in 1:1000){ dat <- data.frame(x = sample(0:1, 100, replace = TRUE), y = sample(0:1, 100, replace = TRUE), z = sample(0:1, 100, replace = TRUE)) dat <- data.frame(apply(dat, 2, as.logical)) } ) } d <- data.frame(func_as.logical, func_transform, func_apply, func_mutate, func_mutate_all, func_mutate_each, func_mutate_at, func_mutate_across) d <- gather(d, key = "func", value = "time", func_as.logical, func_transform, func_apply, func_mutate, func_mutate_all, func_mutate_each, func_mutate_at, func_mutate_across) d$func <- factor(d$func, levels = c("func_as.logical", "func_transform", "func_apply", "func_mutate", "func_mutate_all", "func_mutate_at", "func_mutate_across", "func_mutate_each")) ggplot(d, aes(x = func, y = time)) + geom_boxplot() + coord_flip()
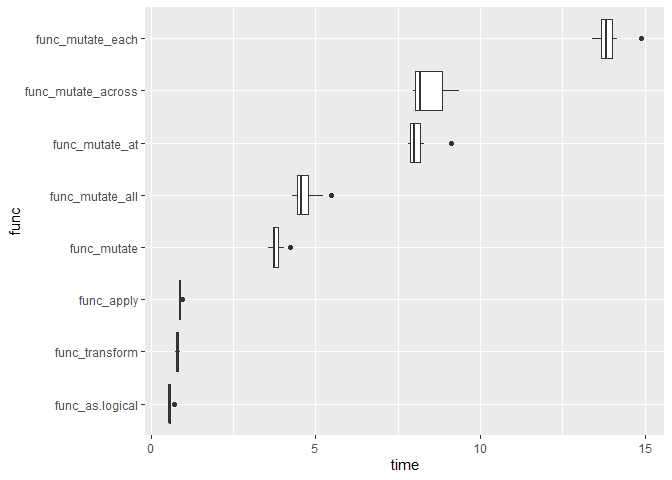
{base}系の方が圧倒的早く、やっぱり圧倒的にmutate_eachが遅い。
sessioninfo
sessionInfo()
## R version 4.0.2 (2020-06-22)
## Platform: x86_64-w64-mingw32/x64 (64-bit)
## Running under: Windows 10 x64 (build 19041)
##
## Matrix products: default
##
## locale:
## [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932
## [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C
## [5] LC_TIME=Japanese_Japan.932
##
## attached base packages:
## [1] stats graphics grDevices utils datasets methods base
##
## other attached packages:
## [1] forcats_0.5.0 stringr_1.4.0 dplyr_1.0.2 purrr_0.3.4
## [5] readr_1.3.1 tidyr_1.1.2 tibble_3.0.3 ggplot2_3.3.2
## [9] tidyverse_1.3.0
##
## loaded via a namespace (and not attached):
## [1] tidyselect_1.1.0 xfun_0.18 haven_2.3.1 colorspace_1.4-1
## [5] vctrs_0.3.4 generics_0.0.2 htmltools_0.5.0 yaml_2.2.1
## [9] blob_1.2.1 rlang_0.4.7 pillar_1.4.6 glue_1.4.2
## [13] withr_2.3.0 DBI_1.1.0 dbplyr_1.4.4 modelr_0.1.8
## [17] readxl_1.3.1 lifecycle_0.2.0 munsell_0.5.0 gtable_0.3.0
## [21] cellranger_1.1.0 rvest_0.3.6 evaluate_0.14 labeling_0.3
## [25] knitr_1.30 fansi_0.4.1 broom_0.7.1 Rcpp_1.0.5
## [29] scales_1.1.1 backports_1.1.10 jsonlite_1.7.1 farver_2.0.3
## [33] fs_1.5.0 hms_0.5.3 digest_0.6.25 stringi_1.5.3
## [37] grid_4.0.2 cli_2.0.2 tools_4.0.2 magrittr_1.5
## [41] crayon_1.3.4 pkgconfig_2.0.3 ellipsis_0.3.1 xml2_1.3.2
## [45] reprex_0.3.0 lubridate_1.7.9 assertthat_0.2.1 rmarkdown_2.4
## [49] httr_1.4.2 rstudioapi_0.11 R6_2.4.1 compiler_4.0.2