Rのggplot2でグラフを描いたときに有意差の記号、*とかそういうのをグラフ上に書こうとしたときの方法について、私的まとめ。 いつもすぐに忘れてググってる。
グラフにannotation描く動機がなければ興味のない話になっている。自分はこうやってるよというのを晒すことでより良い知見にたどり着きたい。
あと必要なときにどこにやったのかわからないRのスクリプト探すより自分のブログをコピペしたほうが早いというのもある。
- ggplotで描いたグラフに有意差のマークを書く方法
- ggplotのgeom_textとgeom_segmentを使って有意差のマークをグラフ上に描く
- ggsignif::geom_signifを使って有意差のマークをグラフ上に描く
- ggpubr::stat_compare_meansを使って有意差のマークをグラフ上に描く
- 雑感
ggplotで描いたグラフに有意差のマークを書く方法
ggplotで書いたグラフに有意差のマークを書く方法は自分の把握している範囲で3つある。
- ggplotで描いたグラフを画像ファイルとして出力したあとに、画像処理用のソフトをつかって画像ファイルに書き込む。
- ggplot上でggplotの関数の
geom_textとgeom_segmentをつかって書き込む。 ggsignif::geom_signifやggpubr::stat_compare_meansなどの別パッケージを使ってggplotで描いた(あるいはggpubrで描いた)グラフに書き込む。
覚えることが少ないのはggplotで描いたグラフを画像ファイルとして出力したあとに、別のソフトをつかって有意差のマークを書き込む方法だが、解像度が微妙に合っていなくて、グラフや有意差のマークのどっちかがボケたり、グラフと線の位置をピッタリ合わせようとすると意外とストレスになる。本稿では触れない。
本稿では、残りの2つの方法について簡素にまとめていく。
個人的な所感としては、データの形がハマればggpubr::stat_compare_meansが楽だが、データがはまらない場合はggsignif::geom_signifが使いやすいかと思う。データがそれぞれのパッケージでうまく処理できない形だと、位置指定をしなければいけないのでちょっと面倒。
ちなみに、ggpubr::stat_compare_meansは内部でggsignif::geom_signifを使っている。
ggplot2の用意しているgeom_textやgeom_segmentを使って描く方法は、要するにグラフ上にせっせと領域指定して罫線ひいているだけなのでだいたい場合で万能。ただし、位置合わせが面倒だったり、コードが長くなる。
ggplotのgeom_textとgeom_segmentを使って有意差のマークをグラフ上に描く
geom_textはグラフ上の任意の箇所に文字(記号)を描く関数で、geom_segmentは始点と終点を指定して罫線を引くggplot2で用意されている関数。
これらをつかってグラフ上にお絵かきしていく。
まずはRの組み込みデータのIRISデータを使って種別のがく片の長さの箱ひげ図を描く
# ggplot2インストールしていなければinstall.packages("ggplot2")でインストールする。 library(ggplot2) ggplot(data = iris, mapping = aes(x = Species, y = Sepal.Length)) + geom_boxplot()

これは見るからに差があるが、一元配置分散分析で確認すると
summary(aov(Sepal.Length~Species, data = iris))
## Df Sum Sq Mean Sq F value Pr(>F)
## Species 2 63.21 31.606 119.3 <2e-16 ***
## Residuals 147 38.96 0.265
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
TukeyHSD(aov(Sepal.Length~Species, data = iris))
## Tukey multiple comparisons of means
## 95% family-wise confidence level
##
## Fit: aov(formula = Sepal.Length ~ Species, data = iris)
##
## $Species
## diff lwr upr p adj
## versicolor-setosa 0.930 0.6862273 1.1737727 0
## virginica-setosa 1.582 1.3382273 1.8257727 0
## virginica-versicolor 0.652 0.4082273 0.8957727 0
とテューキーの多重比較でもすべての群間で有意な差があるということで、ざっとp\<0.001ということで***を各群間に書き込みたくなる。
はじめにsetosaとversicolor間で描いてみる。
ggplot(data = iris, mapping = aes(x = Species, y = Sepal.Length)) + geom_boxplot() + # setosaとverisicolorの2群の間(x = 1.5, y = 7.5)に有意差のマークをつける geom_text(x = 1.5, y = 7.7, label = "***") + # setosa(x = 1の座標)のグラフ上ちょっと上(y = 7.5から7.2に)から縦線をちょっと引いて geom_segment(x = 1, xend = 1, y = 7.5, yend = 7.2) + # setosa(x = 1)からversicolor(x = 2)にさっき引いた直線(y = 7.5)とつながるように線を引いて geom_segment(x = 1, xend = 2, y = 7.5, yend = 7.5) + # Versicolor(x = 2)でグラフの上にちょっと直線をsetosa上に描いたのと同じ位置(y = 7.5から7.2)に描く geom_segment(x = 2, xend = 2, y = 7.5, yend = 7.2)

縦線つけないで横線と米印だけなら2行で済むが、縦線もつけてコを横倒しした形にするとなると4行はコードを書く必要がある。
3群すべてで描くと同様の手順(ただしこれだけではだめなコードを示す)で、
ggplot(data = iris, mapping = aes(x = Species, y = Sepal.Length)) + geom_boxplot() + # setosaとverisicolor間 geom_text(x = 1.5, y = 7.7, label = "***") + geom_segment(x = 1, xend = 1, y = 7.5, yend = 7.2) + geom_segment(x = 1, xend = 2, y = 7.5, yend = 7.5) + geom_segment(x = 2, xend = 2, y = 7.5, yend = 7.2) + # vesicolorとvirginica間 geom_text(x = 2.5, y = 8.5, label = "***") + geom_segment(x = 2, xend = 2, y = 8, yend = 8.3) + geom_segment(x = 2, xend = 3, y = 8.3, yend = 8.3) + geom_segment(x = 3, xend = 3, y = 8, yend = 8.3) + # setosaとvirginica間 geom_text(x = 2, y = 9, label = "***") + geom_segment(x = 1, xend = 1, y = 8.4, yend = 8.7) + geom_segment(x = 1, xend = 3, y = 8.7, yend = 8.7) + geom_segment(x = 3, xend = 3, y = 8.4, yend = 8.7)

となり、ちゃんと描けない。右上のあたりに線が見切れているのでわかるかもしれないが、描画範囲内に有意差のマークが収まっていないから。
ということで、ylimで表示範囲を調整する。
ggplot(data = iris, mapping = aes(x = Species, y = Sepal.Length)) + geom_boxplot() + # setosaとverisicolor間 geom_text(x = 1.5, y = 7.7, label = "***") + geom_segment(x = 1, xend = 1, y = 7.5, yend = 7.2) + geom_segment(x = 1, xend = 2, y = 7.5, yend = 7.5) + geom_segment(x = 2, xend = 2, y = 7.5, yend = 7.2) + # vesicolorとvirginica間 geom_text(x = 2.5, y = 8.5, label = "***") + geom_segment(x = 2, xend = 2, y = 8, yend = 8.3) + geom_segment(x = 2, xend = 3, y = 8.3, yend = 8.3) + geom_segment(x = 3, xend = 3, y = 8, yend = 8.3) + # setosaとvirginica間 geom_text(x = 2, y = 9, label = "***") + geom_segment(x = 1, xend = 1, y = 8.4, yend = 8.7) + geom_segment(x = 1, xend = 3, y = 8.7, yend = 8.7) + geom_segment(x = 3, xend = 3, y = 8.4, yend = 8.7) + # yの範囲を有意差のマークがすべて入るように設定 ylim(c(4.25, 9.1))

でなんとなくいい感じなる。あとは図のサイズなどもろもいじるといい感じになる。
この方法の強みとしては、グラフ上で線や米印をつけたい箇所の座標がわかれば簡単にできる。書きたいところに書きたい長さで書きたいものが簡単にかける。 あとはオプションで色や線の種類も簡単に変えられる。
弱みは入力するものがちょっと多いこと、後述するお絵かき座標用のデータを準備するなどで工夫もできなくない。
あとは、グラフ上の座標を線一本ずつ、米印の位置を入力したり調整したりしないといけない。後述する有意差のマークを付けるためのパッケージをつかった関数を使うと、うまくハマれば位置も簡単に変数名で指定できるし、p値を勝手に計算してくれる。 自分でお絵描きする方法だと計算しなければいけないというのが一手間だが、関数が計算してくれるのも個人的に一長一短があると思っている(この点は最後に書く)。
あとは、上記の書き方だともともと描画される範囲外にマークを書こうとするとylimで範囲指定しなければいけないけど、少しあとで書くaes()で描く方法だと範囲指定がいらない。
今回のIRISデータみたいに位置がわかりやすいと簡単だが、ちょっと複雑だとやりにくい。例えばRの組み込みデータセットのToothGrowthで、ビタミンCの投与量の箱ひげ図を投与方法で層化したグラフを描くと、
ggplot(ToothGrowth, aes(x = factor(dose), y = len, fill = supp)) + geom_boxplot()

となる。
着目する視点次第だけれど今回は微調整が面倒な例として上げたいので、同一投与量内での投与方法での効果の差に注目したときに、
with(ToothGrowth, t.test(len[dose == 0.5 & supp == "OJ"], len[dose == 0.5 & supp == "VC"]))
##
## Welch Two Sample t-test
##
## data: len[dose == 0.5 & supp == "OJ"] and len[dose == 0.5 & supp == "VC"]
## t = 3.1697, df = 14.969, p-value = 0.006359
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## 1.719057 8.780943
## sample estimates:
## mean of x mean of y
## 13.23 7.98
with(ToothGrowth, t.test(len[dose == 1 & supp == "OJ"], len[dose == 1 & supp == "VC"]))
##
## Welch Two Sample t-test
##
## data: len[dose == 1 & supp == "OJ"] and len[dose == 1 & supp == "VC"]
## t = 4.0328, df = 15.358, p-value = 0.001038
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## 2.802148 9.057852
## sample estimates:
## mean of x mean of y
## 22.70 16.77
with(ToothGrowth, t.test(len[dose == 2 & supp == "OJ"], len[dose == 2 & supp == "VC"]))
##
## Welch Two Sample t-test
##
## data: len[dose == 2 & supp == "OJ"] and len[dose == 2 & supp == "VC"]
## t = -0.046136, df = 14.04, p-value = 0.9639
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## -3.79807 3.63807
## sample estimates:
## mean of x mean of y
## 26.06 26.14
となるので、doseが0.5と1では有意な差があるが、2では有意な差がないということでIRIS同様にコを横倒しにしたのに米印を書きたいと思う。
このときに、dose0.5のOJ側のx座標は微妙な位置にあるということ。書きながら微調整していくしか無い。
例としては
ggplot(ToothGrowth, aes(x = factor(dose), y = len, fill = supp)) + geom_boxplot() + # dose0.5 geom_text(x = 1, y = 25, label = "**") + geom_segment(x = 0.81, xend = 0.81, y = 22, yend = 24) + geom_segment(x = 0.81, xend = 1.19, y = 24, yend = 24) + geom_segment(x = 1.19, xend = 1.19, y = 22, yend = 24) + # dose1 geom_text(x = 2, y = 31, label = "**") + geom_segment(x = 1.81, xend = 1.81, y = 28, yend = 30) + geom_segment(x = 1.81, xend = 2.19, y = 30, yend = 30) + geom_segment(x = 2.19, xend = 2.19, y = 28, yend = 30)

となる、x座標が微妙なところになっているので微調整しながら妥協点を見つけていく。一箇所見つければあとは対称な位置にあったりするので簡単だけど。
あとはこれの色やテーマや諸々を調整するといい感じの図になる。
ちなみに、ggplotで図を書くときにggplot()のところにデータやエステティックを書く派と、グラフの形を指定したときに書く派がいると思うが、上記までの書き方だと描けない(次に解決策を書く)。
ggplot() + geom_boxplot(aes(x = Species, y = Sepal.Length), iris) + geom_text(x = 1.5, y = 7.7, label = "***") + geom_segment(x = 1, xend = 1, y = 7.5, yend = 7.2) + geom_segment(x = 1, xend = 2, y = 7.5, yend = 7.5) + geom_segment(x = 2, xend = 2, y = 7.5, yend = 7.2)

geom_textとgeom_segmentのそれぞれでエステティックを書く必要がある。aes()の中身はaes書かなかった上のときと同じでOK。
ggplot() + geom_boxplot(aes(x = Species, y = Sepal.Length), iris) + geom_text(aes(x = 1.5, y = 7.7, label = "***")) + geom_segment(aes(x = 1, xend = 1, y = 7.5, yend = 7.2)) + geom_segment(aes(x = 1, xend = 2, y = 7.5, yend = 7.5)) + geom_segment(aes(x = 2, xend = 2, y = 7.5, yend = 7.2))

この方法だと、範囲指定しなくてもエステティックの要素でグラフの描画範囲をいい塩梅に調整してくれるので、
ggplot() + geom_boxplot(aes(x = Species, y = Sepal.Length), iris) + # setosaとverisicolor間 geom_text(aes(x = 1.5, y = 7.7, label = "***")) + geom_segment(aes(x = 1, xend = 1, y = 7.5, yend = 7.2)) + geom_segment(aes(x = 1, xend = 2, y = 7.5, yend = 7.5)) + geom_segment(aes(x = 2, xend = 2, y = 7.5, yend = 7.2)) + # vesicolorとvirginica間 geom_text(aes(x = 2.5, y = 8.5, label = "***")) + geom_segment(aes(x = 2, xend = 2, y = 8, yend = 8.3)) + geom_segment(aes(x = 2, xend = 3, y = 8.3, yend = 8.3)) + geom_segment(aes(x = 3, xend = 3, y = 8, yend = 8.3)) + # setosaとvirginica間 geom_text(aes(x = 2, y = 9, label = "***")) + geom_segment(aes(x = 1, xend = 1, y = 8.4, yend = 8.7)) + geom_segment(aes(x = 1, xend = 3, y = 8.7, yend = 8.7)) + geom_segment(aes(x = 3, xend = 3, y = 8.4, yend = 8.7))

となる。
ただし、毎回aes()と書くのがちょっと手間。
ちなみにエステティックで描いていればグラフの描画範囲が調整されるので、ggplot()内にデータやエステティックを書く派も、
ggplot(iris, aes(x = Species, y = Sepal.Length)) + geom_boxplot() + # setosaとverisicolor間 geom_text(aes(x = 1.5, y = 7.7, label = "***")) + geom_segment(aes(x = 1, xend = 1, y = 7.5, yend = 7.2)) + geom_segment(aes(x = 1, xend = 2, y = 7.5, yend = 7.5)) + geom_segment(aes(x = 2, xend = 2, y = 7.5, yend = 7.2)) + # vesicolorとvirginica間 geom_text(aes(x = 2.5, y = 8.5, label = "***")) + geom_segment(aes(x = 2, xend = 2, y = 8, yend = 8.3)) + geom_segment(aes(x = 2, xend = 3, y = 8.3, yend = 8.3)) + geom_segment(aes(x = 3, xend = 3, y = 8, yend = 8.3)) + # setosaとvirginica間 geom_text(aes(x = 2, y = 9, label = "***")) + geom_segment(aes(x = 1, xend = 1, y = 8.4, yend = 8.7)) + geom_segment(aes(x = 1, xend = 3, y = 8.7, yend = 8.7)) + geom_segment(aes(x = 3, xend = 3, y = 8.4, yend = 8.7))

でylim()を使わずとも描画範囲を調子してくれる。しかし、打鍵量はaes()を4回を群の数だけ多く書くのでylim()で範囲指定したほうが書く分量は少ない。
エステティックで書く方法の旨味は範囲を自動調整してくれることよりも、座標をデータフレームで与えて描いてもらえることにあると思う。いままで、お絵描きするための座標をそれぞれでベタ打ちしていたが、座標用のデータを作ってそれを参照して作ることもできる。これならば、思った図にならなかっときに直す箇所がベタ打ちしているときよりも少なく、かつ直し忘れが少なくなるかもしれない。まずは、setosaとversicolorの2群間のみに印をつけてみる。
d.setosa <- data.frame(x = 1, xend = 2, y = 7.2, yend = 7.5, label = "***", label.x = 1.5, label.y = 7.7) ggplot(iris, aes(x = Species, y = Sepal.Length)) + geom_boxplot() + geom_text(aes(x = label.x, y = label.y, label = label), d.setosa) + geom_segment(aes(x = x, xend = x, y = y , yend = yend), d.setosa) + geom_segment(aes(x = x, xend = xend, y = yend, yend = yend), d.setosa) + geom_segment(aes(x = xend, xend = xend, y = y, yend = yend), d.setosa)

この方式なら、3群間にしてもgeom_textやgeom_segmentの入力回数も減らせるし、コードもスッキリする。
d.signif <- data.frame(x = c(1, 2, 1), xend = c(2, 3, 3), y = c(7.2, 8, 8.4), yend = c(7.5, 8.3, 8.7), label = c("***", "***", "***"), label.x = c(1.5, 2.5, 2), label.y = c(7.7, 8.5, 9)) ggplot(iris, aes(x = Species, y = Sepal.Length)) + geom_boxplot() + geom_text(aes(x = label.x, y = label.y, label = label), d.signif) + geom_segment(aes(x = x, xend = x, y = y , yend = yend), d.signif) + geom_segment(aes(x = x, xend = xend, y = yend, yend = yend), d.signif) + geom_segment(aes(x = xend, xend = xend, y = y, yend = yend), d.signif)

ということスッキリかけるようになった。
このデータフレームを使う方法は次のように、fillじゃなくてfacet_gridで特定の要因で層化する(パネルを並べるイメージ?)ようなグラフのときに更に生きてくる。具体例として、
ggplot(ToothGrowth, aes(x = supp, y = len)) + geom_boxplot() + facet_grid(cols = vars(dose))

というありがちな感じの、グラフでdose0.5で**をつけようと思ったときに、次のようにするとうまく行かない。
ggplot(ToothGrowth, aes(x = supp, y = len)) + geom_boxplot() + facet_grid(cols = vars(dose)) + geom_text(aes(x = 1.5, y = 31, label = "**")) + geom_segment(aes(x = 1, xend = 1, y = 28, yend = 30)) + geom_segment(aes(x = 1, xend = 2, y = 30, yend = 30)) + geom_segment(aes(x = 2, xend = 2, y = 28, yend = 30))

なんでそうなってしまうかというと、パネルごとにx座標1と2が存在するので、それぞれのパネルごとに同じ位置に描画されてしまう。みんな同じだったらうまーって感じなのだが、今回はdose2で有意な差がなかったので困る。さらにそもそも位置も悪い。
そこでデータを与えてエステティックを描く手法が生きてくる。データにdoseを入れてパネルを選択するようにするとうまくいく。
d.signif <- data.frame(x = c(1, 1), xend = c(2, 2), y = c(28, 30), yend = c(30, 32), label = c("**", "**"), label.x = c(1.5, 1.5), label.y = c(31, 33), dose = c(0.5, 1.0)) ggplot(ToothGrowth, aes(x = supp, y = len)) + geom_boxplot() + facet_grid(cols = vars(dose)) + geom_text(aes(x = label.x, y = label.y, label = label), d.signif) + geom_segment(aes(x = x, xend = x, y = y , yend = yend), d.signif) + geom_segment(aes(x = x, xend = xend, y = yend, yend = yend), d.signif) + geom_segment(aes(x = xend, xend = xend, y = y, yend = yend), d.signif)

他のパッケージの用意している関数を使う場合ではggplot2の仕様が変更になって使えなくなる可能性があるけれど(そうそうないと思うけど)、ggplot2が自前で準備している関数なので仕様変更で使えなくなるという心配がほぼないのもいいところ。
ggsignif::geom_signifを使って有意差のマークをグラフ上に描く
ggsignifはggplotで描いたグラフに有意差のマークを書くためのgeom_signif関数を提供するパッケージ。
github上に開発版があるが、CRANの安定版と変わらない。Githubを見ると今後機能追加はしていかない方針の様子。
install.packages("ggsignif")
# ggplotとggsignifのパッケージを読み込む library(ggplot2) library(ggsignif)
前の説と同じIRISデータでとりあえずversicolorと、virginicaの2群間に有意差の米印をつけてみる。
ggplot(data = iris, mapping = aes(x = Species, y = Sepal.Length)) + geom_boxplot() + # geom_signifで印をつける geom_signif(comparisons = list(c("versicolor", "virginica")), map_signif_level=TRUE)

位置は指定しなければグラフの上となる。線の位置もいい塩梅に、グラフの描画範囲もいい塩梅に調整してくれる。
geom_signifはcomparisonsの引数で比較したい2群を名前でリストで渡して指定する。map_signif_levelの引数でTRUEとすると米印、FALSE(デフォルト)だとp値が直接数値で描画される。
デフォルトだとp値の計算にはwilcox.testが使われる。testの引数で使いたい2群比較でlistでp.valueを持つ検定方法を指定するとよいので例えばt検定使いたかったら、
ggplot(data = iris, mapping = aes(x = Species, y = Sepal.Length)) + geom_boxplot() + # geom_signifで印をつける geom_signif(comparisons = list(c("versicolor", "virginica")), map_signif_level=TRUE, test = "t.test")

とするといい。
米印だと違いがわからないので、米印じゃないバージョンで2つやってみる。
ggplot(data = iris, mapping = aes(x = Species, y = Sepal.Length)) + geom_boxplot() + geom_signif(comparisons = list(c("versicolor", "virginica")))

ggplot(data = iris, mapping = aes(x = Species, y = Sepal.Length)) + geom_boxplot() + # geom_signifで印をつける geom_signif(comparisons = list(c("versicolor", "virginica")), test = "t.test")

ちゃんと結果が違う。
これはlibrary()で適当な2群間比較の検定を読み込んでいると、その他の検定もつかえる。
例えば、Rの組み込み関数のwilcox.testはタイがあると正しいp値を計算できないということが知られているので、代替案としてexactRankTests::wilcox.exactを使いたかったら
library(exactRankTests)
## Package 'exactRankTests' is no longer under development.
## Please consider using package 'coin' instead.
ggplot(data = iris, mapping = aes(x = Species, y = Sepal.Length)) + geom_boxplot() + # geom_signifで印をつける geom_signif(comparisons = list(c("versicolor", "virginica")), test = "wilcox.exact")

とするといい。今回の例だとタイが無いので普通にやった結果と変わらないけれど、特殊な検定を使いたかったらこの方式で計算するのがいい。library名::関数では残念ながら読めないのでtest
= "exactRankTests::wilcox.exact"と指定してもうまく行かない。
2群間の比較しかできないので多重比較の結果で行けないので微妙なところがあるのは否めない。
annotationの引数で入れる文字(記号)を指定できる。
ggplot(data = iris, mapping = aes(x = Species, y = Sepal.Length)) + geom_boxplot() + # geom_signifで印をつける geom_signif(comparisons = list(c("versicolor", "virginica")), annotations = "+++")

高さはデフォルトだとグラフの上部で決まった位置になっているので、3群を何も設定しないで米印をつけようとすると
ggplot(data = iris, aes(x=Species, y=Sepal.Length)) + geom_boxplot() + geom_signif(comparisons = list( c("setosa", "versicolor"), c("versicolor", "virginica"), c("setosa", "virginica")), map_signif_level=TRUE)

とみんな同じ高さになってしまう。離れたところどうし(4群以上で1-2群、3-4群だけ印をつけるなど)なら問題ないのだけれども、こんな感じでくっついてしまう。
y_positionの引数でそれぞれ高さをかぶらないよう調整するといい。さらに調整した高さで勝手に描画範囲がいい塩梅に調整される。
ggplot(iris, aes(x=Species, y=Sepal.Length)) + geom_boxplot() + # これはデフォルトで入る高さにしておく geom_signif(comparisons = list(c("versicolor", "virginica")), map_signif_level=TRUE) + # これはy7.5の高さにしておく geom_signif(comparisons = list(c("setosa", "versicolor")), map_signif_level = TRUE, y_position = 7.5) + # これはy8.5の高さにしておく geom_signif(comparisons = list(c("setosa", "virginica")), map_signif_level = TRUE, y_position = 8.5)

この書き方だとgeom_signifを複数回書かなければいけないので面倒だが、
ggplot(data = iris, aes(x=Species, y=Sepal.Length)) + geom_boxplot() + geom_signif(comparisons = list( c("setosa", "versicolor"), c("versicolor", "virginica"), c("setosa", "virginica")), map_signif_level=TRUE, y_position = c(7.5, 8.1, 8.5))
とy_psitionにc()で高さのベクトルをcomparisonsの順番に合わせて与えても同じ図が出力される。これならgeom_signifを複数書かなくてもいいが、デフォルトの高さで入れられなくなるので、全て任意の位置を指定しなければいけない。
基本的にはコを左90°に回した形になっているが、tip_lengthでマイナス数値を指定するとコを右90°に回した形にできる。あとは、vjustで米印の位置を調整でき、マイナス数値で上にプラス数値で下につくようになる。この要領で、
ggplot(iris, aes(x=Species, y=Sepal.Length)) + geom_boxplot() + geom_signif(comparisons = list(c("versicolor", "virginica")), map_signif_level=TRUE) + geom_signif(comparisons = list(c("setosa", "versicolor")), map_signif_level = TRUE, y_position = 7.5) + # これを下向きにする geom_signif(comparisons = list(c("setosa", "virginica")), map_signif_level = TRUE, # 位置をy4にする y_position = 4, # 足?的なやつの長さを-0.03にして上向きに線が出るようにする tip_length = -0.03, # 米印の位置を横線より下に出るようにする vjust = 3)

でできる。
ただし線の下に米印をつけるように設定すると、あまり遠くに設定すると描画範囲からはみ出てしまう。あくまで自動調整してくれる描画範囲はもともとデフォルトで描画されるだろう範囲のみとなっている。
必要があればylim()で調整する必要がある。
しかし、fillなどでグラフを層化したときには、項目を選んで選択するということがうまくできなくなる。
ggplot(ToothGrowth, aes(x = factor(dose), y = len, fill = supp)) + geom_boxplot() + geom_signif(comparisons = list(c("OJ", "VC")))
## Warning: Computation failed in `stat_signif()`:
## TRUE/FALSE が必要なところが欠損値です
となり意図した図が描けない(マークが付いていない図しか出力されない)ので、せっせと座標を指定して、計算も自前でやってannonationsの引数で入れてやる。
ggplot(ToothGrowth, aes(x = factor(dose), y = len, fill = supp)) + geom_boxplot() + # これは2つに分けてもいい # annonationsで入れたい文字列を入れる、今回はわかりやすいように米印とプラスでかき分けている geom_signif(annotations = c("**", "++"), # yの位置を調整する。最初の方が上のannonationsの最初の選択したほう、以下同じ。 y_position = c(23, 30), # 有意差のコの字の始点のx座標を指定する。 xmin = c(0.81, 1.81), # 有意差のコの字の終点のx座標を指定する。 xmax = c(1.19, 2.19))

足の長さ的なのも調整できるが、デフォルトでもいい感じなるし、米印の位置も線の始点と終点のちょうど真ん中で勝手に調整してくれるので、ggplot2のgeom_textやgeom_segementでやるよりも入力項目が少なくて済むことが多いと思う。
ちなみにfacet_gridで分けた場合はこうなる。
ggplot(ToothGrowth, aes(x = supp, y = len)) + geom_boxplot() + geom_signif(comparisons = list(c("OJ", "VC")), map_signif_level = TRUE, test = "t.test") + facet_grid(cols = vars(dose))

ちなみにその2として、doseの0.5と1とで比較したいと言う場合にはこうなる。
ggplot(ToothGrowth, aes(x = factor(dose), y = len, fill = supp)) + geom_boxplot() + geom_signif(comparisons = list(c("0.5", "1")), map_signif_level = TRUE, test = "t.test")

他にもコの字のグラフに伸びる足のような線をtip_length長くしたり、いろいろ調整することができるのでオプション駆使すればかなり融通は効く。
ggplot(data = iris, mapping = aes(x = Species, y = Sepal.Length)) + geom_boxplot() + geom_signif(comparisons = list(c("setosa", "versicolor")), map_signif_level=TRUE, # tip_lengthで長さを調整できる、最初に描いたほうが左側、次が右側になる。値を一つだけにすると両方とも同じ長さで調整できる tip_length = c(0.5, 0.2))

しかしそんなに微調整したいんだったら、geom_segmentやgeom_textでお絵描きして調整したらいいんじゃないかとも思わなくもない。
エステティックも使えるけれど、どんどん操作が込み入っていくので今回はここまで。基本的にはgeom_textやgeom_segmentでできるようなことと似ているが、ちょっと違うところもあって微妙に使いづらい気がする、基本的にはgeom_signifで準備された引数で処理していくのがいいと個人的には思う。
ggpubr::stat_compare_meansを使って有意差のマークをグラフ上に描く
ggpubrはggplot2ベースでいい感じのグラフを簡単に記載できるようにした関数群を含むパッケージとなっている。
githubに開発版があるが、CRANに安定版があるので
install.packages("ggpubr")
でインストールする。
# ライブラリで読み込む library(ggplot2) library(ggpubr)
ggpubrでグラフを描く関数はggplot2とはやや記法が異なるが、描いたグラフはggplot2で描いたグラフと同じようにgeom_*で装飾できる。逆に、ggpubrで用意されている装飾用の関数のstat_compare_meansなどはggplot2で描いた図も装飾できる。
ggpubr::stat_compare_meansはggsignif::geom_signifを使っており、ggsignif::geom_signifの拡張的な感じ。依存関係でggpubrをインストールするとggsignifもインストールされ、ggpubrをロードするとgeom_signifを使えるようになるし。
ちなみに、ggpubrは依存関係がggsignifに比べて多いのでそういうのを気にするなら選択肢として微妙かもしれない。
とりあえず、ggplot2で描いたIRISデータで3群それぞれに米印を付ける。
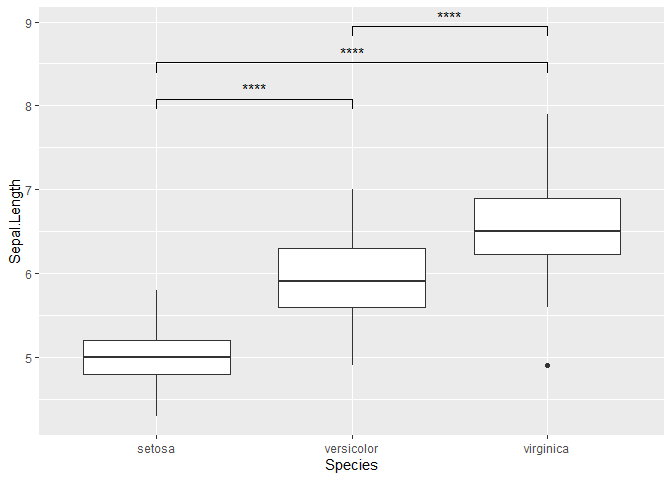
ggplot(iris, aes(x=Species, y=Sepal.Length)) + geom_boxplot() + stat_compare_means(comparisons = list( c("setosa", "versicolor"), c("versicolor", "virginica"), c("setosa", "virginica")), # labelで"p.signif"とすると米印、デフォルトだとp値のままで出力される。 label = "p.signif")

ggsignifではうまく行かなかったコードに近いものでもきれいに描ける。
デフォルトだと米印がggsignifよりも一個多い。
comparisonsのリストは米印をつけたい下からの順番で指定しないと見た目変な描画になってしまう。
ggplot(iris, aes(x=Species, y=Sepal.Length)) + geom_boxplot() + stat_compare_means(comparisons = list( c("setosa", "versicolor"), c("setosa", "virginica"), c("versicolor", "virginica")), label = "p.signif")
デフォルトで用いられるのはwilcox.testなので変えたかったらmethodの引数で他の2群比較の検定を指定するといい。ggsignif同様、libraryで読み込んでいるとその他のライブラリで提供される検定も使える。
例えばt検定を使いたかったら
ggplot(iris, aes(x=Species, y=Sepal.Length)) + geom_boxplot() + stat_compare_means(comparisons = list( c("setosa", "versicolor"), c("versicolor", "virginica"), c("setosa", "virginica")), label = "p.signif", method = "t.test")

とする。
米印はsymnum.argsで調整する。ggsignifだともし出力結果で制御したかったらif文を書かなければいけないと思うのでその点は楽(実用的かどうかは微妙)。
ggplot(iris, aes(x=Species, y=Sepal.Length)) + geom_boxplot() + stat_compare_means(comparisons = list( c("setosa", "versicolor"), c("versicolor", "virginica"), c("setosa", "virginica")), label = "p.signif", symnum.args = list(cutpoints = c(0, 0.001, 0.01, 0.05, 1), symbols = c("***", "**", "*", "ns")))

これで、ggsignifと同じような出力結果に近づく。
そもそも、**じゃなく†使うよ(あるいは他の記号も含めて)、p値0.1から印つけたいよという派も簡単に設定できる。
ggplot(iris, aes(x=Species, y=Sepal.Length)) + geom_boxplot() + stat_compare_means(comparisons = list( c("setosa", "versicolor"), c("versicolor", "virginica"), c("setosa", "virginica")), label = "p.signif", symnum.args = list(cutpoints = c(0, 0.0001, 0.001, 0.01, 0.05, 1), symbols = c("§", "‡", "†", "*", "ns")))

ただし、§なんかは縦長なのでデフォルトだと位置が微妙なので調整が必要。
印の位置はvjustで調整できる。マイナス数値で上に、プラス数値で下に移動する。
ggplot(iris, aes(x=Species, y=Sepal.Length)) + geom_boxplot() + stat_compare_means(comparisons = list( c("setosa", "versicolor"), c("versicolor", "virginica"), c("setosa", "virginica")), label = "p.signif", symnum.args = list(cutpoints = c(0, 0.0001, 0.001, 0.01, 0.05, 1), symbols = c("§", "‡", "†", "*", "ns")), # ほんの僅かに上に上げる。 vjust = -0.2)

あとは基本的にはできることは、ggsignifとよく似ている。
面白いのは対応のある検定を指定できる点。組み込みデータのsleepデータで対応のあるデータをプロットして線でつないだ箱ひげ図を描いてみる。ggplotで描くのがちょっと手がかかるのでggpubrのggpairdで描く。
ggpaired(data = sleep, x = "group", y = "extra", line.color = "gray", xlab = "group", ylab = "extra")

簡単にそれっぽいのが描ける。 ここで、
with(sleep, t.test(extra[group == 1], extra[group == 2]))
##
## Welch Two Sample t-test
##
## data: extra[group == 1] and extra[group == 2]
## t = -1.8608, df = 17.776, p-value = 0.07939
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## -3.3654832 0.2054832
## sample estimates:
## mean of x mean of y
## 0.75 2.33
with(sleep, t.test(extra[group == 1], extra[group == 2], paired = TRUE))
##
## Paired t-test
##
## data: extra[group == 1] and extra[group == 2]
## t = -4.0621, df = 9, p-value = 0.002833
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## -2.4598858 -0.7001142
## sample estimates:
## mean of the differences
## -1.58
となるので、対応のあるt検定ではちゃんと有意な差があるので、そのことを米印をつけたいが、
ggpaired(data = sleep, x = "group", y = "extra", line.color = "gray", xlab = "group", ylab = "extra") + stat_compare_means(comparisons = list(c("1", "2")), label = "p.signif", method = "t.test")

で有意差なしとなってしまう。そこでpariedをTRUEにすると対応のある検定をしてくれる。
ggpaired(data = sleep, x = "group", y = "extra", line.color = "gray", xlab = "group", ylab = "extra") + stat_compare_means(comparisons = list(c("1", "2")), label = "p.signif", method = "t.test", paired = TRUE)

雑感
p値を計算してくれる機能を持つ有意差の米印マークをつけてくれる関数geom_signifやstat_compare_meansについて、その関数が計算できる範囲の検定でうまくはまっているときはうれしいし(たとえば、純粋に2群比較だったり、sleepデータで対応のある検定できるときなど)気持ちがいいが、IRISデータの例ように本来多重比較すべきものも単に2群比較になってしまうというあまりうまくない展開になってしまってくる。
たまたま米印上は同じ結果になっていると図としての成果物としては問題ないのかもしれないけれど、個人的には危うさをはらんでいるなと思う。
最終成果物として、もうこれ以降絶対変更しないというものだったらいいけど、後でデータが変わったりしたときに多重比較してないがゆえなどの不適切な米印がつきかねないなと思っている。
基本的に米印を付ける段階なんて言うのは最後に検定結果を報告する段階にあると思うけど、膨大な量じゃなければgeom_signifやstat_compare_meansが用意している検定結果からマークをつけるのではなく、人力で米印を決めるか、判定間違いやつけ忘れを防ぐためにも自動化したいのであれば少し手間だけれど別途if文で米印のlabelsを決めてやった方が安全だと思う。
関係のない雑感として、ggplot()だと引数の一番最初がdataで、mappingが次だけど、geom_*だとmappingの方が先なので明示しない場合にggplot()と同じように書くとaestheticおかしいよとエラーがでるのが地味にやりにくい。
ほかの関数的にも、dataがaestheticのあとなのは妥当なんだけど、普段ggplot()にいろいろ書いている派なので混乱する。
ggplot()はggplot()でパイプでデータを受けとる設計なのかもしれないのでこれはこれで妥当なんだろうなぁと思った。
あとは極めてどうでもいい雑感としてstat_compare_meansはデフォルトがwilcox.testなのでmeanじゃなくmedianじゃないか(それも正確にはどうなのという話もあるが)、そもそもmeanとかいらなかったのでは、あえてつけたくてどっちもいいよというならお茶を濁す程度にmとかしておけばと思った。
関数名はパッケージ作者の専権事項だし、こんな機能を使おうと思うユーザーが勘違いして平均値比較の検定(t.testがデフォルト)と思いこむこともそうなのでどうでもいいけど。